对于CNV检测而言,全外显子组测序是否足以取代基因芯片?
拷贝数变异(CNV)是个体基因组片段拷贝数差异的一种遗传变异,对医学和进化具有重要意义。据估计,3-14% 的发育障碍患者携带有致病性拷贝数变异,具体比率取决于检测方法、所选择的患者群体。
目前数据库中收录的致病性 CNV 大部分是长度比较长的 CNV,个人认为这主要是由于两个方面:
一、更大的 CNV 包括更多的编码基因,对疾病的影响更大;
二、之前的检测手段比如基因芯片(CMA)和 CNVSEQ 只能检测大于 100 KB 以上的 CNV,几百 bp 到几十 KB 一直是检测的盲区,既然是盲区数据库当然也就没有收录。
最近几年 WES 已成为临床常规检测方案,分析 CNV 的算法已经非常成熟,那么全外显子在 CNV 检测方面能够取代基因芯片吗?
最近发表在 medRxiv 网站的一篇重量级文章也许能够为我们找到部分答案:
该文章介绍了DDD(Deciphering Developmental Disorders)项目对 5197 例患者使用全外显子分析的 CNV 与高分辨率和低分辨率的基因芯片结果进行了对比,WES 采用的是 Agilent SureSelect v3 或 v5,高分辨率芯片采用的是专为DDD项目单独定制的,每个外显子包括 5 个探针,可准确检测单外显子水平CNV,低分辨率芯片是 60K (CytoSure Constitutional v3) 或 180K (Agilent CGH ISCA v2)。
为了准确检测 WES CNV,文章中使用了 CANOES、CLAMMS、CoNVex 和 XHMM 算法,然后使用 random forest 机器学习方法集成了这些算法结果,解决了它们的断裂点位置,通过与父母的结果比较确定了它们的遗传状态。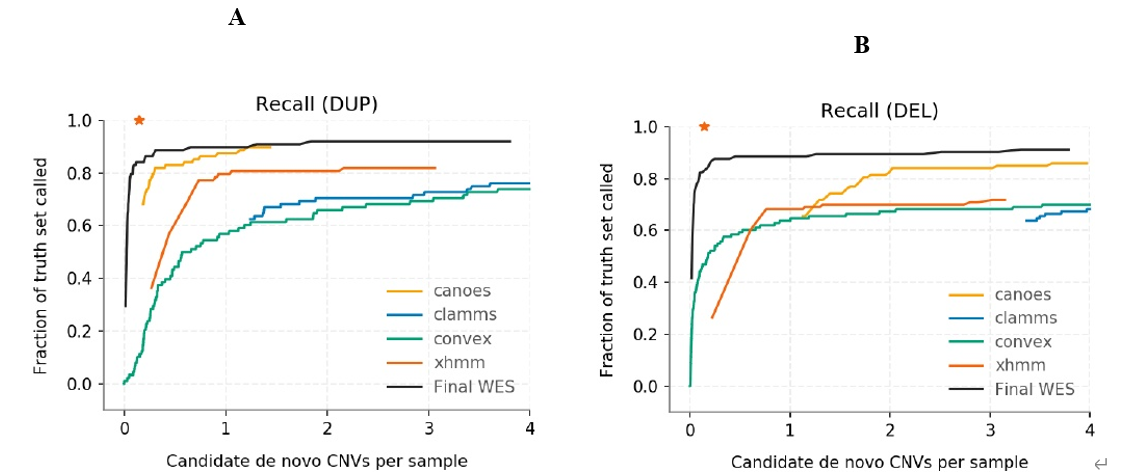
图:ROC曲线显示了各个全外显子算法和我们最终的组合算法在不同质量截断下对拷贝数变异(CNVs)的敏感性
将单个和组合的 WES-based 拷贝数变算法结果与 276 个临床验证的致病性拷贝数变异的真阳性数据集进行比较,发现所有单独算法的准确性远低于组合的 random forest 组合算法(图 1)。整合的 random forest 算法在这个严格的过滤阈值下预测的重复和缺失的新发拷贝数变异的敏感性均为 84%。
接下来评估了对这些个体中已验证的 276 个致病性 CNV 的整体敏感性(主要来自高分辨率的 CMA),组合算法识别出了 246 个(89%)这些 CNV。对于大型CNV(即> 10 个外显子),基于 WES 的结果至少与外显子高分辨率的CMA一样敏感,过滤后的基于 WES 的分析结果达到了 98% 的敏感性,而外显子高分辨率的 CMA 在相同阈值下敏感性仅为 92%。
总的来说,WES 漏掉的大多数致病性 CNV(18/30,60%)与一个(9/30)或零(9/30)个外显子区域重叠,因此无法被发现(补充资料 S12)。虽然外显子分辨率的 CMA 或基于WES的结果中漏掉的大多数致病性 CNV 具有较少的探针数量(<10 个),但其中一些较大的 CNV(WES 漏掉的 30 个中的 4 个)由于被算法分割为小的几个 CNV 而被质控过滤掉。相比之下,外显子高分辨率的 CMA 也识别出了 246 个(89%)经临床验证的 276 个致病性 CNV。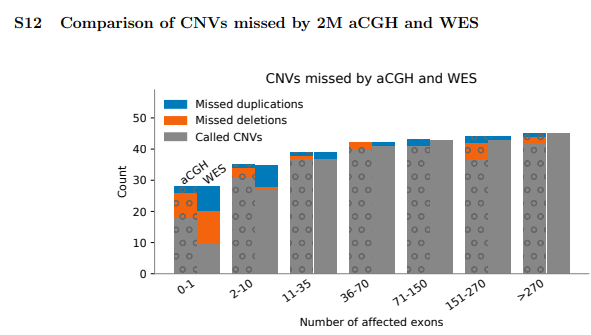
从上面的结果中我们不难看出,即使是对比高分辨率的外显子 CMA,WES 检测 CNV 的性能也不逞多让,而相比金标准遗漏的 CNV 主要是三个方面:
一、单外显子,这主要是因为 WES 本身的算法问题,常规的算法都不去包括这部分 CNV,因为假阳性太高,游侠建议可以使用 DECoN 算法结合可视化外显子作图来排除假阳性;
二、不包括编码区的致病性 CNV,这是 WES 天然的检测盲区,除非 WES 特殊设计 CNV 骨架,这方面游侠经历过 2 次深刻的体会,第一次是性发育异常疾病 DSD 致病基因 SOX9 上游有一段非编码调控区,第二次是性连锁耳聋 POU3F4 基因上游的调控区;
三、算法检测出不连续的小 CNV,这主要是 WES 检测的 CNV 信号不稳定,把一段长的CNV分割为几个短的小 CNV,所以游侠提醒遗传分析人员要注意此种特殊情况而不遗漏。
在招募参加 DDD 研究的 9,859 名拥有 trio 外显子组测序数据的患者中,有 7,182 人(73%)之前曾接受过低分辨率 CMA 进行大型致病性 CNV 的临床检测。在那些之前接受过低分辨率 CMA 测试的患者中,观察到 2.6% 的患者通过高分辨率 CMA 或基于外显子组测序检测到额外的致病性CNV。在之前没有接受低分辨率 CMA 的 27% 参与者中,确定了 280 名参与者(3%),在这个群体中,我们观察到来自 de novo CNVs 的更高诊断阳性率为 5.0%。比较这两个群体的 CNV 诊断阳性率表明,WES 可以检测到的致病性 CNV 中有 52% 是低分辨率 CMA 无法检测到的。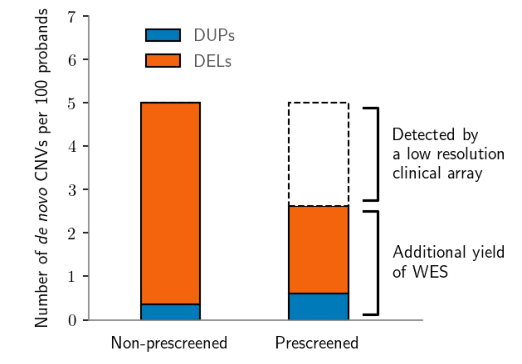
所以,全外显子组测序是否足以取代基因芯片?相信大家心里都有了答案了吧!
SureSelect 临床研究外显子组 V4 由安捷伦与世界知名临床分子遗传学家、美国埃默里遗传学实验室前执行主任 Madhuri Hegde 博士及其团队合作开发,是专业医学洞见和安捷伦十余年捕获探针合成经验的完美结合;2016 年推出的 CRE V2 在就是当时临床研究中覆盖最为全面的人全外显子组产品,CRE V4 则在 CRE V2 基础上结合最新临床需求,进一步在基因内容选择、探针合成与铺设、NGS 湿实验流程、实验室自动化等层面进行了更迭与创新,更加适用于遗传性疾病临床研究。
SureSelect 临床研究外显子组 V4 的核心特点与价值
01 41.1Mb 靶标区域,48.5Mb 探针设计大小,高测序性价比
02 6500+ 疾病相关基因,性染色体加强覆盖;包含深内含子变异,41 个 mini 基因,1500+ 祖源SNP,100+ 药物基因组 SNP,常见致病 CNV 等
03 全面覆盖最新人类蛋白质编码和经优选的非编码区域
04 针对 Bravo 和 Magnis 文库自动化平台优化
05 由权威遗传病专家团队精心挑选和优化
座机
021-58390070
发送您的留言
微信扫码咨询

