安捷伦病原体检测方案与应用案例 - 基因芯片篇
18 世纪,人类通过显微镜首次识别出微生物。自那以后,我们已经了解了微生物如何参与物理环境和有机生物圈的调节,以及它们与单细胞和多细胞生物之间的广泛联系。临床上,如何更快更经济的鉴定致病微生物;生物医学研究中,如何全面分析微生物对人类健康和疾病的影响及重要性,都是当下临床研究人员在不断为之努力的课题。
为了高效鉴定与特定疾病相关的病原体,研究人员应用了包括 PCR 扩增后进行 16s rRNA 测序在内的多种宏基因组工具。然而,病原体种类多样,既包含真菌、细菌,也包括病毒、寄生虫和原生动物,不是用一把锤子就可以解决所有问题的。比如 16s rRNA 测序就不适用于病毒和真核微生物。同样,由于人类宿主 DNA 会产生高背景,对总 DNA 样本进行宏基因组测序可能会导致产率较低;而对于背景菌较多的样本,宏基因组测序数据的解读也更为困难。
为了应对这些挑战,可以采用 DNA 微阵列芯片作为有效的分析平台,快速、轻松且经济地对大量样品进行广泛的微生物筛查。
PathoChip 病原体基因芯片
宾夕法尼亚大学佩雷尔曼医学院的团队利用安捷伦比较基因组杂交 (aCGH) 芯片的设计算法,设计了一款覆盖 6000 多种原核和真核人类病原体的芯片——PathoChip。该芯片的探针覆盖了所有已知和公开的病毒序列、数百种致病菌、真菌、酵母和寄生虫。这款芯片还包含了病原体家族内部特定的,以及病原体家族之间保守序列的探针,因此能够检测新的微生物和/菌株。得益于 aCGH 基因芯片的技术原理,PathoChip 对病原体的检测灵敏度不受宿主污染的影响,并且可在单次检测中同时鉴定 RNA 和 DNA 病原体。PathoChip 的检测数据可以通过 GeneSpring GX 软件进行分析。
PathoChip 的设计
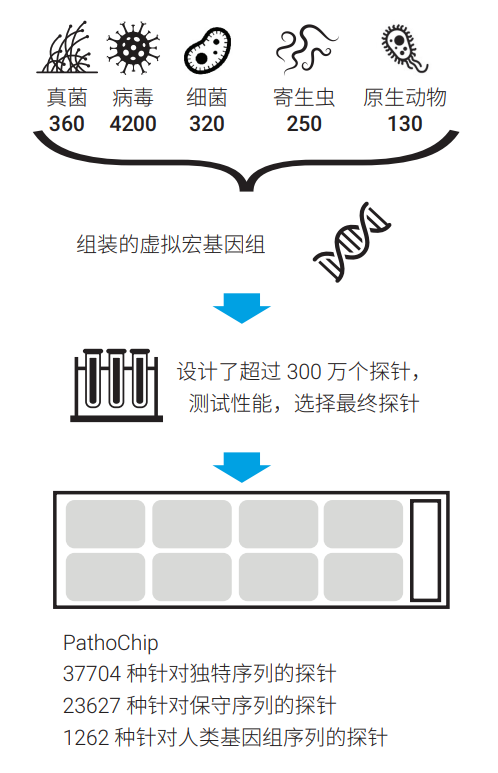
研究人员汇总了 6000 多种原核和真核人类病原体,将由此产生的基因序列组装成一个宏基因组,并将其分成 58 条虚拟“染色体”,每条染色体由大约 500–1000 万个核苷酸组成。使用安捷伦比较基因组杂交芯片 (aCGH) 的设计算法,设计了针对宏基因组中独特区域和保守区域的探针序列[1, 2]。然后对这些序列进行过滤,以降低与人类基因组序列交叉杂交的可能性,从而尽可能降低背景噪音。研究人员最初创建了超过 300 万个探针,通过性能测试,确定了纳入最终设计的探针列表。
座机
021-58390070
发送您的留言
微信扫码咨询

